Nous avons parlé de l’ADN et de l’ARN, il est à présent temps de s’intéresser aux protéines. Cours de rappel : l’ADN est transcrit en ARN qui à son tour est traduit en protéines. Ces protéines ont des rôles très divers en fonction de leur localisation tissulaire et cellulaire, leur capacité à transformer des substrats, leur conformation 3D… La totalité des protéines de notre corps forme le protéome et elle est étudiée grâce à des techniques de protéomique.
Protéomique & vieillissement : comment étudie-t-on le protéome ?
La protéomique relève de l’étude du protéome, qui est l’ensemble des protéines d’une cellule. Dans ces analyses, on peut s’intéresser aux modifications que subit une protéine lors de sa synthèse : sa traduction, les modifications post-traductionnelles (comme l’acétylation, la méthylation…), son repliement (la structure 3D d’une protéine lui permet de fonctionner), son couplage avec d’autres protéines (formation de dimères, trimères ou polymères) ou encore sa dégradation. On rappelle que les protéines peuvent se retrouver sous plusieurs formes :
- la forme primaire est la succession des acides aminés qui constituent la protéine. C’est la séquence protéique. Elle peut être touchée par les modifications post-traductionnelles comme la méthylation ou l’actéylation.
- la forme secondaire est la forme repliée des protéines. C’est la structure que prend la protéine afin d’optimiser sa future fonction. On pourra observer la formation locale de feuillets, d’hélices ou de coudes.
- la structure tertiaire est la forme 3D de la protéine. Une fois la structure secondaire adoptée, il faut à nouveau la replier pour lui donner une conformation dans l’espace. Cela est possible par la formation de liaisons entre différents sites protéiques.
- la structure quaternaire est la forme finale des protéines, mais n’est pas adoptée par toutes les protéines. En effet, elle est le résultat d’une association de plusieurs chaines protéiques pour former des polymères. Certaines protéines fonctionnent sous forme de monomère et s’arrêtent donc à une structure tertiaire.
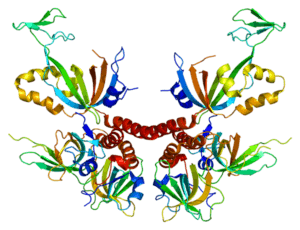
La protéomique permet d’étudier toutes ces structures et d’identifier des cibles thérapeutiques potentielles ou des biomarqueurs de pathologies. Ses applications se diversifient de plus en plus, notamment sur les interactions protéine-ADN, protéine-ARN et protéine-protéine, rejoignant ainsi la génomique, la transcriptomique et la biologie des systèmes.
Préparation des protéines
Afin de pouvoir étudier le protéome, il faut d’abord extraire et séparer les différentes protéines avant de les identifier puis de les quantifier. Pour cela, on part d’un tissu (biopsie, organe, sang…) ou de cellules (en culture notamment) que l’on va détruire afin d’extraire les protéines. Pour ce faire, il existe plusieurs techniques, la plus classique étant l’utilisation d’un produit chimique, en combinaison avec des anti-protéases (des enzymes qui dégradent les protéines et que l’on bloque), pour casser les membranes des cellules et récupérer les protéines présentes à l’intérieur. Cette étape est extrêmement importante car une bonne extraction est garante d’une bonne analyse par la suite. Il est important de noter que, suite à cette étape d’extraction, les protéines se retrouvent sous leur forme primaire, c’est à dire sans repliement ou structure 3D. Pour étudier les formes secondaires et tertiaires des protéines, il existe d’autres techniques, plus complexes, que nous ne détaillerons pas.
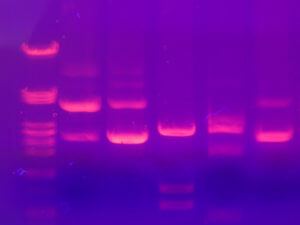
Après extraction, on récupère un extrait protéique, le plus pur possible. Il est alors nécessaire de séparer les protéines selon leurs caractéristiques physico-chimiques. Les protéines ont en effet des tailles différentes, sont plus ou moins chargées en fonction de leur composition, sont plus ou moins acides ou basiques et vont parfois pouvoir se lier à d’autres protéines qu’on appelle des ligands. Pour les séparer, on se base donc sur ces propriétés et on utilise le plus souvent une électrophorèse ou une chromatographie.
L’électrophorèse consiste en un gel placé entre deux électrodes (une positive et une négative) permettant le passage d’un courant. Cette technique va séparer les protéines en fonction de leur taille, les plus petites migrant plus rapidement sur le gel, les plus grosses restant dans le haut du gel. Lorsqu’on rajoute une étape préalable de migration iso-électrique, on peut également séparer les protéines en fonction de leur charge avant de les séparer par leur taille. On peut ainsi obtenir des petits spots sur un gel, correspondant aux protéines.
La chromatographie est quant à elle, une technique totalement différente bien que se basant sur les mêmes propriétés. L’extrait protéique est injecté dans une colonne de chromatographie (un tube contenant des filtres de diverses natures) afin de récupérer uniquement un type de protéine. Dans la colonne, on peut avoir des filtres de taille, qui ne laissent passer que les petites protéines, des filtres de charges, qui ne laissent passer que les protéines chargées positivement par exemple, ou encore des filtres d’affinité, qui contiennent un ligand sur lequel une protéine spécifique va se fixer. Tous ces différents types de colonnes permettent le même type de séparation que les électrophorèses mais ont plus pour but la purification de l’extrait protéique que l’analyse à proprement parler.
Identification et quantification des protéines
Une fois les protéines extraites et purifiées, les outils de protéomique permettent l’identification, l’analyse et la quantification de celles-ci. On distingue notamment la technique de western blot, qui permet de faire migrer des protéines sur un gel d’électrophorèse, de transférer ce résultat sur une membrane puis d’hybrider cette membrane avec un anticorps spécifique de notre protéine d’intérêt. On peut ainsi détecter uniquement une protéine donnée, puis la quantifier grâce à une référence interne au test. La technique du 2D-DIGE (pour « Fluorescence two-dimensional differential gel electrophoresis ») se base sur le même principe [1]. Le problème principal de ces procédés est la limitation à un gel d’électrophorèse, ne rendant pas possible l’analyse de protéines à grande échelle. Des tests permettant l’analyse de plus grandes quantités de protéines existent également, comme la spectrométrie de masse ou les puces à protéines. La première est assez complexe, se basant sur le marquage des protéines grâce à des radio-isotopes : après digestion par des enzymes, on obtient plusieurs fragments protéiques marqués, permettant la quantification et l’identification exacte de toutes les protéines contenues dans l’échantillon [2]. Les puces à protéines sont quant à elles, très similaires aux puces à ADN ou ARN [3].
Que faire des données de protéomique ?
L’application principale de la protéomique reste l’étude des interactions entre protéines dysfonctionnelles et pathologies, ainsi que le développement de biomarqueurs et de cibles thérapeutiques d’une maladie donnée [4]. Une fois les protéines identifiées et quantifiées, on peut en effet lier leur abondance (augmentation ou baisse de synthèse) et leur forme (mutation, modifications…) à un trouble. Tout comme les données de génomique et transcriptomique, les résultats d’une étude de protéomique apportent une meilleure compréhension des pathologies, à un niveau différent et complémentaire des études sur l’ADN et l’ARN [4].
Quelques exemples de modifications protéiques
Modification de la séquence protéique
L’ADN contient l’information permettant à l’ARN d’être traduit en protéines suivant une séquence d’acides aminés spécifiques. Lorsqu’une mutation a lieu dans l’ADN, cela va se répercuter sur la séquence protéique. C’est ce qu’on appelle le code génétique : la combinaison de trois bases d’ADN va donner un codon et il existe actuellement 61 codons différents donnant lieu à 20 acides aminés. Par exemple, un codon TTT (Thymine x3) va donner une phénylalanine (un des 20 acides aminés), alors qu’on codon TCT (Thymine-Cytosine-Thymine) va donner une sérine. Cela peut sembler dérisoire comme différence, mais la structure protéique finale dépend entièrement de sa composition en acides aminés. S’il existe une mutation d’un codon TTT en TCT, alors la phénylalanine sera remplacée par une sérine dans la protéine, créant de potentiels problèmes en terme de repliement, de site actif ou de site de liaison.
Altération des modifications post-traductionnelles
Si la protéine a été correctement synthétisée, elle n’est pas à l’abri d’autres problèmes. Une protéine ne se résume pas à sa séquence, il faut qu’elle soit modifiée pour être fonctionnelle. Ces modifications post-traductionnelles sont très variées et peuvent consister en l’ajout de groupement phosphate (phosphorylation), acétyl (acétylation), méthyl (méthylation), de sucres divers (glycosylation) ou encore d’ubiquitine (ubiquitinylation), cette dernière étant un système de reconnaissance de la cellule permettant l’élimination de la protéine. Toutes ces modifications peuvent être altérées, augmentées ou diminuées, impactant la fonction de la protéine.
Modifications de la structure 3D
La forme finale d’une protéine consiste en son repliement dans l’espace et son éventuelle association avec d’autres protéines. Afin d’effectuer son repliement, il est nécessaire de créer des liaisons entre les différentes parties de la protéine. Ces liaisons sont diverses et peuvent aller de liaisons fortes, comme les liaisons covalentes maintenant nos molécules, à des liaisons faibles, comme les liaisons ioniques, qui se basent sur l’interaction entre des zones chargées positivement et des zones chargées négativement au sein de la protéine.
Quel impact pour notre corps ?
Prenons un exemple simple et connu de tous : la mucoviscidose. C’est une maladie génétique rare touchant les muqueuses et le système d’excrétion de notre corps. Les premiers symptômes sont généralement pulmonaires, avec une accumulation de mucus empêchant une respiration normale, puis s’étendant au foie, à l’intestin, au pancréas voire aux muqueuses génitales, entraînant la mort entre 20 et 40 ans.
Pourquoi parler de cette maladie ? Parce qu’elle est l’exemple même d’un dysfonctionnement protéique. En effet, le gène codant pour la protéine CFTR est muté, entraînant la formation d’une protéine tronquée, incapable de se replier correctement et de se loger dans la membrane des cellules comme elle devrait l’être. Il existe malheureusement beaucoup d’autres exemples de maladies de ce type, dont l’origine n’est pas forcément une mutation génétique.
Applications de la protéomique au vieillissement
Il est évident qu’au-delà de leur utilité pour les maladies rares, les outils de protéomique ont également un intérêt dans la recherche contre le vieillissement. En effet, l’intérêt majeur de ces tests est l’approche à grande échelle et la prise en compte de milliers de données traitées par la suite grâce des techniques bio-informatiques [5]. Cela permet l’identification de nouvelles protéines centrales à la mise en place de pathologies.
Par exemple, une étude récente a disséqué les voies de signalisation menant à l’accumulation de l’amyloide β dans la maladie d’Alzheimer, mettant en évidence de nouvelles hypothèses [6]. Une autre s’est intéressée au vieillissement des reins chez le rat, associant 66 protéines avec un vieillissement ralenti et 73 autres avec un vieillissement accéléré. Cette étude suggère que l’environnement rénal, en particulier vis à vis du stress oxydatif et de la fonction mitochondriale, est de première importance dans le vieillissement rénal et la dégradation de sa fonction [7]. Dans la mitochondrie, une équipe a également mis au point de nouveaux outils permettant de suivre sa fonction au cours du temps [8]. Toujours dans la mitochondrie, des études récentes de protéomique ont identifié les sites d’acétylation, succinylation, glutarylation et malonylation (toutes des modifications post-traductionnelles) et leur régulation par les sirtuines, SIRT3 et SIRT5, des protéines largement impliquées dans le vieillissement [9]. Concernant la sarcopénie liée à l’âge, une équipe a mis en relation le statut oxydatif et la perte musculaire [10]. D’autres études se sont penchées sur la protéostasie, dont la perte est l’une des 9 causes du vieillissement, et son effet sur la longévité [11].
La liste des recherches en cours pourrait continuer encore et encore, tant les espoirs portés par la protéomique sont importants dans la lutte contre le vieillissement, mais nous nous arrêterons à ces quelques exemples. Pour plus d’information sur le protéome et les études actuellement en développement, vous pouvez visiter le site du Human Proteome Project (en anglais) ou celui de l’EBI (en anglais), pour aller plus loin dans votre formation à la protéomique !
Tout notre dossier Approches « omics »
Les approches « omics » : mieux comprendre notre vieillissement
Qu’est-ce qu’il se cache derrière les approches « omics » ? Quand on parle de génomique, transcriptomique ou protéomique, à quoi s’intéresse-t-on ? Un petit guide pour mieux comprendre !
Partie 1 : Parlons peu, parlons génomique !

Difficile de ne pas s’étendre sur la génomique et toutes ses applications. C’est la plus ancienne des approches « omics » mais c’est aussi celle qui est la plus étudiée. Elle a donné naissance au concept d’épigénétique et ouvert la porte à des espoirs jusque là impossibles !
Partie 2 : La transcriptomique, une science en constante évolution.

La découverte des ARN non codants a valu un prix Nobel, c’est dire l’importance de cette discipline. La transcriptomique c’est l’ouverture d’un champ de possibles, la découverte de nouveaux mécanismes et la compréhension progressive de la régulation de la transcription.
Partie 3 : Protéomique, un meli-melo de disciplines
 La protéomique, c’est avant tout une approche multiple, prenant en compte les interactions avec d’autres disciplines, notamment la génomique, et faisant appel à des concepts intercroisés avec l’immunologie, la fonction cellulaire ou la nutrition.
La protéomique, c’est avant tout une approche multiple, prenant en compte les interactions avec d’autres disciplines, notamment la génomique, et faisant appel à des concepts intercroisés avec l’immunologie, la fonction cellulaire ou la nutrition.
Partie 4 : La métabolomique, la dernière-née des « omics »
 « Last but not least » comme diraient nos amis anglophones. La métobolomique, c’est la compréhension de réseaux de régulation complexes et la découverte quotidienne de nouveaux acteurs dans les communications entre nos cellules.
« Last but not least » comme diraient nos amis anglophones. La métobolomique, c’est la compréhension de réseaux de régulation complexes et la découverte quotidienne de nouveaux acteurs dans les communications entre nos cellules.
Références
[1] R Tonge, J Shaw, B Middleton, et al. Validation and development of fluorescence two-dimensional differential gel electrophoresis proteomics technology. Proteomics. 2001;1(3):377–96
[2] RW Nelson, JR Krone, AL Bieber, P Williams, Peter. Mass Spectrometric Immunoassay. Analytical Chemistry. 1995;67(7):1153–1158
[3] DH Wilson, DM Rissin, CW Kan et al. The Simoa HD-1 Analyzer: A Novel Fully Automated Digital Immunoassay Analyzer with Single-Molecule Sensitivity and Multiplexing. J Lab Autom. 2016;21(4):533–47
[4] AD Weston, L Hood. Systems Biology, Proteomics, and the Future of Health Care: Toward Predictive, Preventative, and Personalized Medicine. Journal of Proteome Research. 2004;3(2):179–96
[5] S Rogers, M Girolami, W Kolch, KM Waters, T Liu, B Thrall, HS Wiley. Investigating the correspondence between transcriptomic and proteomic expression profiles using coupled cluster models. Bioinformatics. 2008;24(24):2894–2900
[6] Yu L, Petyuk VA, Gaiteri C, Mostafavi S, et al. Targeted brain proteomics uncover multiple pathways to Alzheimer’s dementia. Ann Neurol. 2018. doi: 10.1002/ana.25266. [Epub ahead of print]
[7] Li D, Zhao D, Zhang W, Ma Q et al. Identification of proteins potentially associated with renal aging in rats. Aging (Albany NY). 2018. doi: 10.18632/aging.101460. [Epub ahead of print]
[8] Cortassa S, Sollott SJ, Aon MA. Computational Modeling of Mitochondrial Function from a Systems Biology Perspective. Methods Mol Biol. 2018;1782:249-265
[9] Carrico C, Meyer JG, He W, Gibson BW, Verdin E. The Mitochondrial Acylome Emerges: Proteomics, Regulation by Sirtuins, and Metabolic and Disease Implications. Cell Metab. 2018;27(3):497-512
[10] Smith NT, Soriano-Arroquia A, Goljanek-Whysall K, Jackson MJ, McDonagh B. Redox responses are preserved across muscle fibres with differential susceptibility to aging. Journal of Proteomics. 2018;177:112-123
[11] Basisty N, Meyer JG, Schilling B. Protein Turnover in Aging and Longevity. Proteomics. 2018 Mar;18(5-6):e1700108
Dr. Marion Tible

Author/Reviewer
Auteure/Relectrice
Marion Tible has a PhD in cellular biology and physiopathology. Formerly a researcher in thematics varying from cardiology to neurodegenerative diseases, she is now part of Long Long Life team and is involved in scientific writing and anti-aging research.
More about the Long Long Life team
Marion Tible est docteur en biologie cellulaire et physiopathologie. Ancienne chercheuse dans des thématiques oscillant de la cardiologie aux maladies neurodégénératives, elle est aujourd’hui impliquée au sein de Long Long Life pour la rédaction scientifique et la recherche contre le vieillissement.
En savoir plus sur l’équipe de Long Long Life
Dr Guilhem Velvé Casquillas

Author/Reviewer
Auteur/Relecteur
Physics PhD, CEO NBIC Valley, CEO Long Long Life, CEO Elvesys Microfluidic Innovation Center
More about the Long Long Life team
Docteur en physique, CEO NBIC Valley, CEO Long Long Life, CEO Elvesys Microfluidic Innovation Center
En savoir plus sur l’équipe de Long Long Life